La falta de conocimiento sobre estas proteínas y la importancia de este descubrimiento se debe a que no había ningún procedimiento tecnológico para identificarlas este tipo de pequeñas proteínas.
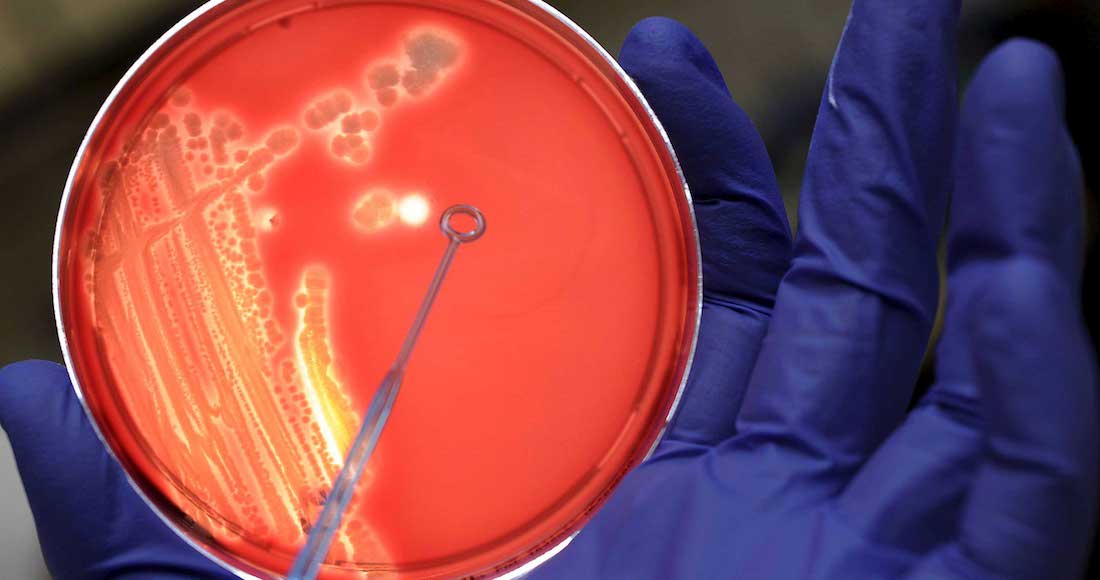
Barcelona, 22 febrero (EFE).- Una nueva técnica permite identificar y ver cómo funcionan pequeñas proteínas presentes en las bacterias, lo que supone un avance para conseguir nuevos antibióticos que superen las resistencias a estos fármacos.
El estudio, publicado hoy en la revista Molecular Systems Biology, ha sido dirigido por Luis Serrano, jefe del grupo de Diseño de Sistemas Biológicos, y ha tenido como investigadora principal a María Lluch-Senar, ambos del Centro de Regulación Genómica (CRG) de Barcelona.
Hasta el momento, no había ningún método efectivo para detectar todas las pequeñas proteínas en un organismo y además saber qué función ejercen; tan solo se conocía que había una gran cantidad de ellas y que estaban relacionadas con el sistema inmune, explicó a Efe Lluch-Senar.
Asimismo, estas moléculas «están muy relacionadas con la salud de los humanos, sobre todo con la flora intestinal, y tienen mucha relación con el sistema de defensa de nuestro cuerpo», dijo el investigador español.
Recientemente, la comunidad científica consiguió aclarar la utilidad concreta de estas partículas pequeñas: los insectos, animales, bacterias e incluso humanos secretamos estas proteínas cuando nuestro organismo detecta una infección y sirven para combatirla.
Además, las bacterias las usan para combatir a otras bacterias, comunicarse entre ellas e incluso también con el huésped, que es el propio ser humano.
Por lo tanto, este hallazgo abre la puerta al posible desarrollo y creación de nuevos antibióticos, que puedan derrotar a las bacterias cada vez más fuertes e inmunes a los medicamentos ya existentes.

La Organización Mundial de la Salud (OMS) ha alertado de este fenómeno, que ha calificado de «una de las mayores amenazas para la salud mundial, la seguridad alimentaria y el desarrollo, que puede afectar a cualquier persona, independientemente de su edad y país de residencia».
«Con esta técnica informática que hemos desarrollado podemos saber todas las pequeñas proteínas en una bacteria y, posteriormente, podría ser extrapolado a los humanos y descubrir algunas proteínas que seguramente estamos obviando», señaló Lluch-Senar.
La falta de conocimiento sobre estas proteínas y, a su vez, la importancia de este descubrimiento se debe a que este tipo de proteínas son extremadamente pequeñas y no había ningún procedimiento tecnológico para identificarlas.
El primer autor del trabajo y estudiante de doctorado del CRG, Samuel Miravet-Verde, señaló que su objetivo principal «era determinar el número y variedad de funciones, para poder presentar todas las proteínas que no se conocían hasta ahora».
De hecho, en una de las bacterias estudiadas que tiene el nombre científico de Mycoplasma pneumoniae y es una de las que causa enfermedades como la pulmonía, la investigación ha determinado que se ignoraban 10 de cada 100 pequeñas proteínas que tiene, a pesar de su reducido tamaño.
En total, el CGR ha concluido que no se tenían en cuenta hasta un 16 por ciento de estas proteínas que forman parte del genoma de todos los seres vivos.
Es por ello que los científicos creen que si se consigue aplicar esta técnica a humanos podría revelarse que hay muchas de estas pequeñas proteínas que aún no conocemos.
Una vez conocida cuál es la tarea de estas moléculas, se podrá detectar cuáles podrán servir como antibióticos, lo que es esperanzador, teniendo en cuenta que actualmente cada vez hay más casos en que estos medicamentos no funcionan porque las bacterias se vuelven resistente a ellos.
La investigación se ha llevado a cabo analizando 109 muestras genéticas de bacterias, lo que ha permitido que la técnica que han desarrollado sea universal y se pueda aplicar a otras especies bacterianas.